MINI RECENZJA
Placówka przeciw rakowi: uniwersalne markery wyłącznie nowotworowe
Chengchen Qian1, Xiaolong Zou2, Wei Li1,3, Yinshan Li4, Wenqiang Yu5
1Shanghai Epiprobe Biotechnology Co., Ltd, Szanghaj 200233, Chiny;2 Oddział Chirurgii Ogólnej, Pierwszy Szpital Stowarzyszony Uniwersytetu Medycznego w Harbin, Harbin 150001, Chiny;3Shandong Epiprobe Medical Laboratory Co., Ltd, Heze 274108, Chiny;4 Szpital Ludowy Regionu Autonomicznego Ningxia Hui, Uniwersytet Medyczny Ningxia, Yinchuan 750002, Chiny;5Szanghajskie Centrum Kliniczne Zdrowia Publicznego i Oddział Chirurgii Ogólnej, Szpital Huashan i Instytut Przerzutów Nowotworowych i Laboratorium Epigenetyki RNA, Instytuty Nauk Biomedycznych, Szanghajska Szkoła Medyczna, Uniwersytet Fudan, Szanghaj 200032, Chiny
ABSTRAKCYJNY
Rak jest główną przyczyną zgonów na całym świecie.Wczesne wykrycie raka może obniżyć śmiertelność w przypadku wszystkich typów nowotworów;jednakże w przypadku większości typów nowotworów brakuje skutecznych biomarkerów wczesnego wykrywania.Metylacja DNA zawsze była głównym przedmiotem zainteresowania, ponieważ metylacja DNA zwykle zachodzi przed innymi wykrywalnymi zmianami genetycznymi.Podczas badania wspólnych cech raka przy użyciu nowatorskiego sekwencjonowania pozycjonowania przewodników na potrzeby metylacji DNA, pojawiła się seria uniwersalnych markerów nowotworowych (UCOM) jako silnych kandydatów do skutecznego i dokładnego wczesnego wykrywania raka.Chociaż wartość kliniczna obecnych biomarkerów nowotworowych jest zmniejszona ze względu na niską czułość i/lub niską swoistość, unikalne cechy UCOM zapewniają klinicznie znaczące wyniki.Walidacja potencjału klinicznego UCOM w leczeniu raka płuc, szyjki macicy, endometrium i nabłonka dróg moczowych dodatkowo wspiera zastosowanie UCOM w wielu typach nowotworów i różnych scenariuszach klinicznych.W rzeczywistości zastosowania UCOM są obecnie przedmiotem aktywnych badań i dalszej oceny w zakresie wczesnego wykrywania raka, diagnostyki pomocniczej, skuteczności leczenia i monitorowania nawrotów.Kolejnym ważnym tematem do zbadania są mechanizmy molekularne, za pomocą których UCOM wykrywają nowotwory.Zastosowanie UCOM w rzeczywistych scenariuszach również wymaga wdrożenia i udoskonalenia.
SŁOWA KLUCZOWE
Wykrywanie raka;badania przesiewowe w kierunku raka;metylacja DNA;epigenetyka nowotworów;biomarkery nowotworowe
Dlaczego pilnie potrzebujemy nowego biomarkery?
Po ponad stu latach walki z rakiem, rak nadal stanowi najbardziej śmiertelne zagrożenie biologiczne dla ludzkości.Rak pozostaje globalnym problemem zdrowotnym z 19,3 milionami nowych przypadków i prawie 10 milionami zgonów według szacunków w 2020 r.1. Szacuje się, że w 2020 r. w Chinach zdiagnozowano około 4,6 miliona nowych przypadków raka, co stanowi 23,7% nowych przypadków nowotworów na całym świecie według GLOBOCAN1.Ponadto w 2020 r. w Chinach nowotwory przypisano około 3 milionom zgonów, co stanowiło 30% wszystkich zgonów spowodowanych nowotworami na świecie1.Statystyki te wskazują, że Chiny zajmują pierwsze miejsce pod względem zachorowalności i umieralności na nowotwory.Co więcej, wskaźnik 5-letniego przeżycia nowotworowego wynosi 40,5% i jest 1,5 razy niższy niż wskaźnik 5-letniego przeżycia w Stanach Zjednoczonych2,3.Stosunkowo niższe wskaźniki przeżywalności i wyższe wskaźniki śmiertelności w Chinach niż w krajach o wyższych wskaźnikach rozwoju społecznego sugerują, że pilnie potrzebny jest skuteczny i opłacalny system zapobiegania nowotworom i nadzoru nad nimi.Wczesne wykrycie nowotworu jest jednym z najważniejszych elementów systemu opieki zdrowotnej.Wczesne wykrycie nowotworu może poprawić rokowanie i przeżycie na wczesnym etapie w przypadku prawie wszystkich typów nowotworów4.Skuteczne strategie badań przesiewowych doprowadziły do znacznego spadku częstości występowania i umieralności na raka szyjki macicy, piersi, jelita grubego i prostaty.
Jednak wczesne wykrycie raka nie jest zadaniem łatwym.Największymi przeszkodami w tym procesie zawsze było badanie biologii i prognozowanie wczesnego nowotworu, identyfikacja i walidacja wiarygodnych biomarkerów wczesnego wykrywania oraz opracowywanie dostępnych i dokładnych technologii wczesnego wykrywania4.Precyzyjne wykrycie nowotworu pozwala na odróżnienie zmian łagodnych od złośliwych, co pozwala uniknąć niepotrzebnych zabiegów i ułatwia dalsze leczenie choroby.Obecne strategie wczesnego wykrywania obejmują biopsję endoskopową, obrazowanie medyczne, cytologię, testy immunologiczne i testy biomarkerów5-7.Biopsje endoskopowe, inwazyjne i kosztowne, niosą ze sobą duże obciążenie jako główna procedura medyczna wymagająca zaangażowania profesjonalnego personelu.Podobnie jak cytologia, obie metody badań przesiewowych zależą od personelu medycznego i opierają się na osobistej ocenie, a ich skuteczność jest daleka od ideału8.Natomiast testy immunologiczne są wysoce niedokładne, biorąc pod uwagę wysoki odsetek wyników fałszywie dodatnich.Obrazowanie medyczne jako taktyka badań przesiewowych wymaga drogiego sprzętu i wyspecjalizowanych techników.Dlatego obrazowanie medyczne jest niezwykle ograniczone ze względu na niską dostępność.Z tych wszystkich powodów biomarkery wydają się lepszą opcją wczesnego wykrywania raka.
Korespondencja do: Yinshan Li i Wenqiang Yu
Email: liyinshan@nxrmyy.com and wenqiangyu@fudan.edu.cn
Identyfikator ORCID: https://orcid.org/0009-0005-3340-6802 i
https://orcid.org/0000-0001-9920-1133
Otrzymano 22 sierpnia 2023 r.;zaakceptowano 12 października 2023 r.;
opublikowano w Internecie 28 listopada 2023 r.
Dostępne na www.cancerbiomed.org
©2023 Biologia i medycyna nowotworów.Creative Commons
Uznanie autorstwa — licencja międzynarodowa 4.0 na licencji niekomercyjnej
Biomarkery są obecnie klasyfikowane jako białka, markery mutacji DNA, markery epigenetyczne, nieprawidłowości chromosomalne, markery RNA pochodzące bezpośrednio z nowotworów lub fragmenty nowotworów uzyskane pośrednio z płynów ustrojowych.Markery białkowe są najpowszechniej stosowanymi biomarkerami w badaniach przesiewowych i diagnostyce nowotworów.Biomarkery białkowe, jako biomarkery przesiewowe, są ograniczone przez tendencję do występowania zmian łagodnych, co prowadzi do nadmiernej diagnozy i nadmiernego leczenia, jak donoszono w przypadku α-fetoproteiny i antygenu specyficznego dla prostaty (PSA)9,10.Markery RNA obejmują wzorce ekspresji genetycznej i inne niekodujące markery RNA. Kombinację markerów RNA ekspresji genetycznej można wykryć za pomocą próbek moczu, których czułość była daleka od zadowalającej (60%) w przypadku guzów pierwotnych, a których wykrycie może podlegać wpływowi łatwej degradacji RNA w normalnym środowisku11.Zarówno markery genetyczne, jak i epigenetyczne stoją przed problemem częstości występowania nowotworów i ograniczenia do typów nowotworów.
Metylacja DNA jest mocnym kandydatem na biomarker wczesnego wykrywania, odkąd Feinberg po raz pierwszy powiązał ją z rakiem w 1983 r. 12. Aberracje w metylacji DNA obserwuje się we wszystkich stadiach raka, już w stadium przednowotworowym.Nieprawidłowa hipermetylacja DNA zwykle ma miejsce na wyspach CpG w promotorach genów, aby przeciwdziałać supresorom nowotworu13,14.Badania sugerują również, że nieprawidłowa hipermetylacja DNA uczestniczy w regulacji w górę regulatorów rozwoju15.Dolina metylacji DNA, która jest powszechnie kojarzona z regulatorami rozwoju i hipermetylowanymi nowotworami, może przełączyć tryb ekspresji genów na bardziej stabilny tryb zależny od metylacji DNA i zmniejszyć połączenie z metylowanym histonem H3K27me3 i powiązanymi białkami polikombowymi16,17.
Spośród dużej liczby opublikowanych markerów metylacji DNA kilka z sukcesem zadebiutowało na rynku;jednakże obecnie skomercjalizowane markery metylacji DNA i panele diagnostyczne nie uwolniły jeszcze w pełni potencjału wczesnego wykrywania raka z wielu powodów18.Chociaż biomarkery te wykazują przeważnie akceptowalną wydajność na podstawie informacji z baz danych, w świecie rzeczywistym te biomarkery zwykle działają gorzej ze względu na fakt, że próbki ze świata rzeczywistego są często bardziej złożone i nie tak reprezentatywne jak te wybrane w bazach danych.Wykazano, że wczesne wykrywanie metylacji wielu nowotworów w oparciu o sekwencjonowanie nowej generacji ma czułość zaledwie 16,8% i 40,4% w przypadku nowotworów w stadium I i II19.Testy wczesnego wykrywania wymagają większej stabilności i dokładniejszych biomarkerów.
Wykrywanie uniwersalnego markera nowotworowego (UCOM) za pomocą sekwencjonowania pozycjonowania przewodnika (GPS)
Pomimo dziesięcioleci badań nad rakiem nie udało się osiągnąć zadowalającej profilaktyki i leczenia.Potrzebne są nowe metodologie, które umożliwią naukowcom dokładną ocenę nowotworu.W ciągu ostatnich 23 lat 6 cech charakterystycznych raka, takich jak unikanie apoptozy, inwazja tkanek i przerzuty itp., zostało rozszerzonych do 14 poprzez uwzględnienie takich cech, jak niezmutowane przeprogramowanie epigenetyczne i polimorficzne mikrobiomy20,21.W miarę ujawniania coraz większej liczby szczegółów dotyczących raka, do badań nad rakiem pojawia się coraz więcej perspektyw.Badania nad rakiem stopniowo weszły w nową erę w dwóch kierunkach (wspólność i indywidualność).Wraz z rozwojem onkologii precyzyjnej w ostatnich latach, badania nad nowotworami skupiają się na zindywidualizowanej terapii celowanej i heterogeniczności nowotworu22.Dlatego też niedawno zidentyfikowane biomarkery nowotworowe skupiają się głównie na określonych typach nowotworów, takich jak rak szyjki macicy PAX623 i BMP3 w przypadku raka jelita grubego24.Skuteczność tych biomarkerów specyficznych dla typów nowotworów jest różna, ale nadal nie jest możliwe, aby osoby podatne poddały się badaniom przesiewowym w kierunku wszystkich nowotworów jednocześnie ze względu na ograniczenia w pobieraniu próbek biologicznych i wysokie koszty.Idealnie byłoby, gdybyśmy mogli zidentyfikować pojedynczy, solidny biomarker, który jest skuteczny w przypadku wszystkich typów nowotworów na wczesnym etapie.
Aby osiągnąć taki idealny cel, należy wybrać lepszego kandydata na biomarker z listy potencjalnych typów biomarkerów.Wiadomo, że aberracje metylacji DNA, spośród wszystkich profili genetycznych i epigenetycznych, są powiązane z rakiem i są jednymi z najwcześniejszych, jeśli nie pierwszymi nieprawidłowościami związanymi z nowotworem, które występują chronologicznie.Badanie metylacji DNA rozpoczęło się wcześnie, ale było utrudnione przez brak metod badawczych.Spośród 28 milionów potencjalnych metylowanych miejsc CpG w genomie należy wykryć możliwą do opanowania liczbę i dopasować do genomu, aby lepiej zrozumieć powstawanie nowotworów.Sekwencjonowanie wodorosiarczynem całego genomu (WGBS), uważane za złoty standard sekwencjonowania metylacji DNA, może pokryć jedynie 50% C w komórkach nowotworowych ze względu na charakter obróbki wodorosiarczynem, który rozbija fragmenty DNA i zmniejsza złożoność genomu podczas transformacja Cs-do-Ts25.Inne metody, takie jak chipy 450 tys., obejmują jedynie 1,6% metylacji genomu.Na podstawie danych 450 tys. panel wykrywający metylację DNA ma czułość 35,4% dla 6 typów nowotworów w I stadium26.Ograniczenia typów nowotworów, słaba wydajność i szum generowany przez metody wykrywania w procesie analitycznym stały się największymi przeszkodami dla paneli wykrywających raka.
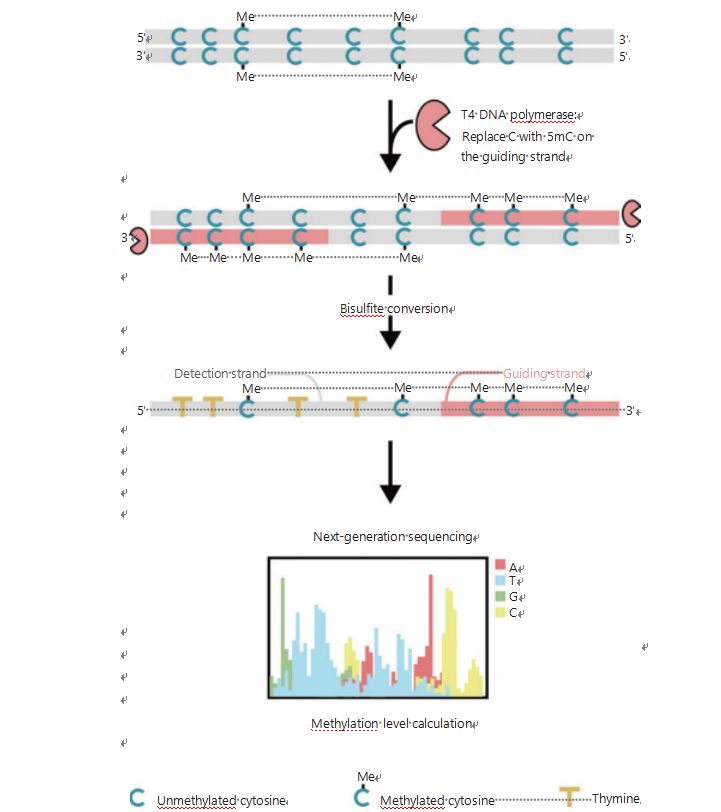
Aby lepiej zbadać wzorce epigenetyczne komórek podczas powstawania nowotworów i przerzutów, opracowaliśmy unikalny GPS do wykrywania metylacji DNA w całym genomie, który obejmuje do 96% miejsc CpG w 0,4 miliarda odczytów25.GPS to dwustronna metoda sekwencjonowania wykorzystująca koniec 3' fragmentu DNA nie ulegających konwersji metylocytozyn po obróbce wodorosiarczynem, która pomaga w wyrównaniu obliczeń metylacji DNA końca 5' poprzez sekwencjonowanie końców par (ryc. 1)25.Nić prowadząca metylocytozyna, działająca jako nić matrycowa, pomaga w dopasowaniu regionu o wysokiej GC, co pozwala odzyskać najbardziej porzucone dane sekwencjonowania w tradycyjnym WGBS.Funkcja GPS o dużym zasięgu zapewnia ogromną ilość informacji o metylacji DNA, co pozwala nam badać profile metylacji nowotworów ze znacznie większą rozdzielczością w wcześniej niedostatecznie zbadanych regionach.
GPS zapewnia nam potężne narzędzie do badania jednorodności nowotworu, co może znacznie uprościć badania nad nowotworami i potencjalnie znaleźć uniwersalne wyjaśnienie powstawania nowotworów i przerzutów.Analizując dane GPS dotyczące linii komórek nowotworowych, często spotykano się z unikalnym zjawiskiem.W wielu typach próbek nowotworu było wiele obszarów, które wydawały się być nieprawidłowo hipermetylowane.To nieoczekiwane odkrycie zostało następnie zweryfikowane i służyło jako UCOM.Przeanalizowano ponad 7000 próbek z 17 typów nowotworów znajdujących się w bazie danych The Cancer Genome Atlas (TCGA), spośród których zidentyfikowaliśmy pierwszy UCOM, HIST1H4F, gen powiązany z histonami, który ulega hipermetylacji we wszystkich typach nowotworów27.Następnie odnaleziono i zweryfikowano serię UCOM w bazie danych TCGA, bazie danych Gene Expression Omnibus (GEO) oraz w rzeczywistych próbkach klinicznych.Jak dotąd, HIST1H4F, PCDHGB7 i SIX6 zostały znalezione i zatwierdzone jako UCOM.Nieoczekiwane odkrycie UCOM oferuje skuteczną odpowiedź na potrzebę wczesnego wykrywania raka.UCOM zapewniają rozwiązanie do wykrywania jednego markera wielu nowotworów.
Charakterystyka UCOM
Po walidacji wykazano, że UCOM wykazują cztery główne cechy, dzięki którym UCOM przewyższają skuteczność obecnych biomarkerów (ryc. 2).
Wyjątkowy dla nowotworu złośliwego
UCOM są charakterystyczne dla zmian nowotworowych lub przednowotworowych i nie mają na nie wpływu normalne zmiany fizjologiczne.Niektóre z obecnych markerów nowotworowych, które są szeroko stosowane we wczesnym wykrywaniu i/lub badaniach przesiewowych, prowadzą do nadmiernej diagnozy.Podwyższone poziomy PSA – klinicznie akredytowane narzędzie przesiewowe – wykrywa się także w łagodnych schorzeniach, takich jak przerost prostaty i zapalenie gruczołu krokowego10.Nadmierne rozpoznawanie i wynikające z niego nadmierne leczenie prowadzą do pogorszenia jakości życia z powodu powikłań jelitowych, moczowych i seksualnych28.Inne oparte na białkach biomarkery szeroko stosowane w warunkach klinicznych, takie jak CA-125, nie przyniosły żadnych znaczących korzyści, powodując jednocześnie nadmierne rozpoznawanie i nadmierne leczenie29.Wysoka specyficzność UCOM w leczeniu nowotworów pozwala uniknąć tych niedociągnięć.UCOM, PCDHGB7, skutecznie odróżnia płaskonabłonkowe zmiany śródnabłonkowe wysokiego stopnia (HSIL) i raka szyjki macicy od normalnych próbek i śródnabłonkowe zmiany płaskonabłonkowe niskiego stopnia (LSIL), podczas gdy większość innych biomarkerów pozwala jedynie na odróżnienie raka szyjki macicy od normalnych próbek30.Chociaż PCDHGB7 nie wykrywa znaczących różnic między prawidłowym endometrium a rozrostem endometrium, wykrywa się istotne różnice między prawidłowym endometrium a atypowym rozrostem, a jeszcze większe różnice wykrywa się między prawidłowym endometrium a rakiem endometrium (EC) na podstawie PCDHGB731.UCOM są unikalne dla zmian złośliwych w bazach danych i próbkach klinicznych.Z perspektywy pacjenta unikalne UCOM zmniejszają próg zrozumienia złożonych wskazań różnych niestabilnych biomarkerów o słabym działaniu i odpowiadający im niepokój podczas procesu oceny.Z punktu widzenia klinicysty unikalne UCOM pozwalają na odróżnienie nowotworów złośliwych od zmian łagodnych, co ułatwia selekcję pacjentów oraz ogranicza niepotrzebne procedury medyczne i nadmierne leczenie.Dlatego unikalne UCOM zmniejszają redundancję systemu medycznego, łagodzą problemy systemu i udostępniają potrzebującym więcej zasobów medycznych.
Rysunek 1 Schemat działania GPS przy wykrywaniu metylacji DNA25.Szara linia: wejściowa sekwencja DNA;czerwona linia: DNA traktowane polimerazą DNA T4, zastępując cytozynę 5′-metylocytozyną na 3′ końcu wejścia;niebieski C z Me: metylowana cytozyna;niebieski C: niemetylowana cytozyna;żółty T: tymina25.
Wszystko albo nic
UCOM są obecne tylko w komórkach nowotworowych i są wykrywane stabilnie w prawie wszystkich komórkach nowotworowych.Potwierdzono, że HIST1H4F jest hipermetylowany w prawie wszystkich typach nowotworów, ale nie w normalnych próbkach27.Podobnie wykazano, że PCDHGB7 i SIX6 są hipermetylowane we wszystkich próbkach nowotworu, ale nie w normalnych próbkach30-32.Ta unikalna cecha znacznie poprawia wydajność UCOM w odniesieniu do granicy wykrywalności i czułości.W próbkach można różnicować zaledwie 2% komórek nowotworowych, co czyni UCOM znacznie czulszym biomarkerem niż większość istniejących biomarkerów30. Mutacje KRAS, będące biomarkerem stosowanym do wykrywania raka jelita grubego, występują jedynie w około 36% przypadków raka jelita grubego. co sugeruje słaby potencjał diagnostyczny33.Niska częstość występowania mutacji KRAS w raku jelita grubego ogranicza KRAS w połączeniu z innymi biomarkerami.W rzeczywistości kombinacja biomarkerów może początkowo wydawać się obiecująca, ale nie zawsze daje zadowalający wynik, wykazując jednocześnie znacznie większy szum w analizie wykrywania i zwykle wiąże się z bardziej skomplikowanymi procedurami eksperymentalnymi.Natomiast PCDHGB7 i inne UCOM występują we wszystkich nowotworach.UCOM wykrywają składniki nowotworowe w różnych typach próbek nowotworów z najwyższą precyzją, eliminując jednocześnie złożone procesy analizy redukcji szumów.Wykrycie raka w dużej próbce nie jest trudne, ale wykrycie raka w małej próbce jest niezwykle trudne.UCOM są w stanie wykryć niewielkie ilości nowotworów.
Rysunek 2 Charakterystyka UCOM.
Wykrywanie nowotworu poprzedzającego zmiany patologiczne
UCOM można wykryć w fazie przednowotworowej, przed wystąpieniem zmian patologicznych.Jako biomarkery epigenetyczne, nieprawidłowości UCOM pojawiają się na wcześniejszym etapie niż nieprawidłowości fenotypowe i są wykrywalne podczas powstawania nowotworu, progresji i przerzutów34,35.Czułość UCOM z biegiem czasu zwiększa skuteczność UCOM w wykrywaniu raka we wczesnym stadium i zmian przednowotworowych.Wykrycie wczesnego nowotworu na podstawie biopsji i cytologii może być trudne nawet dla najbardziej doświadczonych patologów.Donoszono, że pojedyncza biopsja pobrana za pomocą kolposkopii dała wynik pozytywny w 60,6% próbek HSIL+.Aby zwiększyć czułość, w przypadku wielu zmian konieczne są dodatkowe biopsje36.Natomiast UCOM, PCDHGB7, ma czułość 82% dla próbek HSIL+, przewyższającą czułość biopsji i większości biomarkerów30.Marker metylacji, FAM19A4, ma czułość 69% dla CIN2+, która jest podobna do cytologii, ale nie pozwala na odróżnienie CIN1 od normalnych próbek37.Wykazano, że UCOM są znacznie czulszym biomarkerem wczesnego wykrywania.W porównaniu z patologami opartymi na doświadczeniu, UCOM mają lepszą czułość wykrywania nowotworów we wczesnych stadiach, co z kolei przyczynia się do poprawy rokowań i przeżycia w przypadku raka30.Dodatkowo UCOM oferują platformę detekcyjną dostępną dla obszarów, w których brakuje doświadczonych patologów, i znacznie poprawiającą skuteczność wykrywania.Dzięki jednolitym procedurom pobierania próbek i wykrywania wykrywanie UCOM zapewnia stabilne i łatwe do interpretacji wyniki, które lepiej pasują do protokołu badań przesiewowych wymagającego mniejszej liczby profesjonalnego personelu i zasobów medycznych.
Łatwe do wykrycia
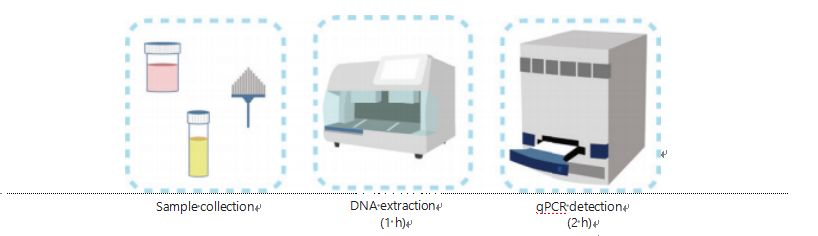
Obecne metody wykrywania metylacji DNA są skomplikowane i czasochłonne.Większość metod wymaga transformacji wodorosiarczynem, co powoduje utratę jakości próbki i prawdopodobnie daje niestabilne i niedokładne wyniki.Słaba powtarzalność spowodowana leczeniem wodorosiarczynem potencjalnie prowadzi do zamieszania wśród lekarzy i pacjentów oraz w dalszym stopniu zakłóca kontrolę i/lub strategie leczenia.Dlatego dalej zmodyfikowaliśmy metodę wykrywania UCOM, aby uniknąć problematycznej obróbki próbek wodorosiarczynem, dostosować się do wymagań zastosowań klinicznych i zwiększyć dostępność.Opracowaliśmy nowatorską metodę wykorzystującą wrażliwe na metylację enzymy restrykcyjne w połączeniu z fluorescencyjną ilościową PCR w czasie rzeczywistym (Me-qPCR) w celu ilościowego określenia stanu metylacji UCOM w ciągu 3 godzin przy użyciu łatwych w obsłudze procedur (ryc. 3).Me-qPCR może obsługiwać wiele typów próbek, np. kliniczną zbiórkę płynów ustrojowych i próbki moczu pobrane samodzielnie.Zebrane próbki kliniczne można przetwarzać, przechowywać i łatwo przystąpić do wykrywania za pomocą standaryzowanej i zautomatyzowanej ekstrakcji DNA.Wyekstrahowany DNA można następnie bezpośrednio zastosować na platformie Me-qPCR w celu przeprowadzenia reakcji w jednym naczyniu i uzyskania wyników ilościowych.Po prostej analizie wyników przy użyciu modeli diagnostycznych dopasowanych i zweryfikowanych dla określonych typów nowotworów, ostateczne określenie wyników wykrywania UCOM jest interpretowane i przedstawiane jako wartość półilościowa.Platforma Me-qPCR przewyższa tradycyjne pirosekwencjonowanie wodorosiarczynem w detekcji UCOM, jednocześnie oszczędzając 3 godziny konwersji wodorosiarczynem, zgodnie z protokołem zestawu EZ DNA Methylation-Gold.Innowacyjna platforma wykrywania metylacji sprawia, że wykrywanie UCOM jest stabilniejsze, dokładniejsze i bardziej dostępne30.
Rysunek 3 Proces wykrywania UCOM.Typy próbek obejmują profesjonalnie pobraną próbkę BALF, szczoteczkę Pap i/lub mocz pobrany samodzielnie.Proces ekstrakcji DNA można dostosować do automatycznego ekstraktora, którego produkt można bezpośrednio wykryć metodą qPCR.
Zastosowanie UCOM
Rak płuc
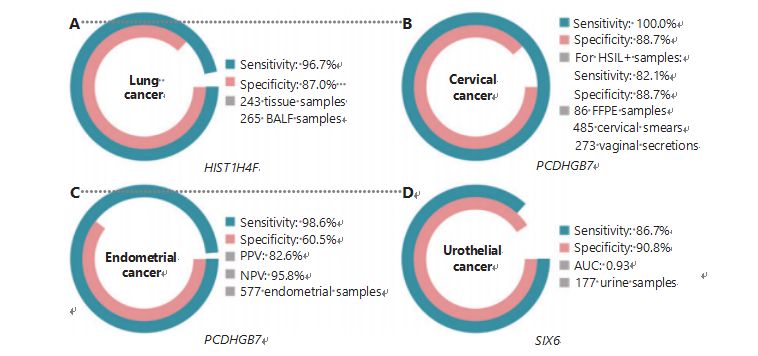
Rak płuc jest drugim najczęściej diagnozowanym i najbardziej śmiertelnym nowotworem na świecie, odpowiadając za 11,4% nowych przypadków i 18,0% nowych zgonów1.Spośród wszystkich rozpoznań 85% to niedrobnokomórkowy rak płuc (NSCLC), a 15% to drobnokomórkowy rak płuc (SCLC), który charakteryzuje się wyższym stopniem złośliwości38.Skanowanie niskodawkową tomografią komputerową (LDCT) jest obecnie zalecaną metodą badań przesiewowych w kierunku raka płuc i wykazano, że poprawia wczesne wykrywanie i zmniejsza śmiertelność6;jednakże ze względu na niską swoistość i słabą dostępność LDCT nie okazała się jeszcze zadowalającą metodą przesiewową, podobnie jak inne powszechnie stosowane markery nowotworowe, takie jak CEA39.Koszty i możliwość pominięcia i błędnego rozpoznania strategii badań przesiewowych LDCT utrudniają postęp w promocji badań przesiewowych w kierunku raka płuc40.HIST1H4F, UCOM, ma ogromny potencjał jako biomarker wczesnego wykrywania w próbkach płynu oskrzelowo-pęcherzykowego (BALF)27.HIST1H4F ulega hipermetylacji w gruczolakoraku płuc i raku płaskonabłonkowym płuc, ze swoistością wykrywania 96,7% i czułością 87,0% (ryc. 4A) oraz wyjątkową skutecznością w przypadku nowotworów w stadium I27.HIST1H4F ma swoistość 96,5% i czułość 85,4% dla NSCLC oraz odpowiednio 96,5% i 95,7% dla SCLC27.Dodatkowo próbki ośmiu innych typów nowotworów, w tym raka trzustki i jelita grubego, potwierdziły, że HIST1H4F jest hipermetylowany we wszystkich ośmiu typach27.
Rak szyjki macicy
Rak szyjki macicy był czwartym najczęściej diagnozowanym nowotworem i czwartą najczęstszą przyczyną zgonów z powodu nowotworów u kobiet w 2020 r., odpowiadając za 3,1% nowych przypadków i 3,4% zgonów związanych z nowotworami na całym świecie1.Aby wyeliminować raka szyjki macicy do 2030 r., zgodnie z propozycją WHO, konieczne jest wczesne wykrycie raka szyjki macicy.W przypadku wykrycia we wczesnym stadium, wskaźnik przeżycia 5-letniego w przypadku inwazyjnego raka szyjki macicy sięga 92%.41Wytyczne American Cancer Society (ACS) sugerują wykonanie badań cytologicznych szyjki macicy, podstawowych badań HPV lub dodatkowych badań przesiewowych42.Cytologia szyjki macicy jest metodą inwazyjną i umożliwia wykrycie jedynie 63,5% przypadków CIN2+37.
Natomiast PCDHGB7 radzi sobie znacznie lepiej z wymazami cytologicznymi i wydzieliną z pochwy i może skutecznie odróżnić HSIL od LSIL na bardzo wczesnym etapie.Sam PCDHGB7 ma czułość 100,0% i swoistość 88,7% w przypadku raka szyjki macicy (ryc. 4B) oraz czułość i swoistość 82,1% i 88,7% w przypadku próbek HSIL+30.PCDHGB7 charakteryzuje się także czułością 90,9% i swoistością 90,4% w przypadku próbek wydzieliny z pochwy w kierunku raka szyjki macicy, które są znacznie łatwiejsze do pobrania30.W połączeniu z testem HPV wysokiego ryzyka (hr) lub testem cytologicznym Thinprep (TCT), PCDHGB7 ma zwiększoną czułość 95,7% i swoistość 96,2%, znacznie przewyższając test hrHPV (20,3%), TCT (51,2% ) i dwa łącznie (57,8%) w przypadku raka szyjki macicy30.Wykazano również, że PCDHGB7 jest hipermetylowany w 17 typach nowotworów z bazy danych TCGA, co wskazuje na jego przydatność w rodzinie UCOM30.
Rycina 4 UCOM zostały potwierdzone w badaniach klinicznych na dużą skalę w czterech typach nowotworów.A. Wydajność HIST1H4F, UCOM, w wykrywaniu raka płuc w 508 próbkach.B. Wydajność PCDHGB7, UCOM, w wykrywaniu raka szyjki macicy w 844 próbkach.C. Działanie PCDHGB7, UCOM, w wykrywaniu raka endometrium w 577 próbkach szczoteczek Pap i Tao endometrium.D. Działanie SIX6, UCOM, w wykrywaniu raka nabłonka dróg moczowych w 177 próbkach.
EC
EC jest jednym z najczęstszych nowotworów układu rozrodczego kobiet na świecie, z szacunkową liczbą 4,2 miliona nowych przypadków i 1% zgonów związanych z nowotworami rocznie1.Przy pomyślnej diagnozie na wczesnym etapie EC jest uleczalna, a wskaźnik przeżycia 5-letniego wynosi 95% w przypadku raka w I stadium.Pacjentki, u których występują objawy, takie jak nieprawidłowe krwawienie z macicy, poddawane są okresowej ocenie klinicznej i poddawane inwazyjnej i bolesnej biopsji, mimo że tylko u 5–10% ostatecznie rozwija się EC43.Przezpochwowe USG, jako powszechna metoda wykrywania, jest wysoce zawodne ze względu na niemożność odróżnienia zmian łagodnych od złośliwych endometrium oraz wysoki odsetek wyników fałszywie dodatnich44.
Przeprowadzono równoległe porównanie CA-125 w surowicy, szeroko stosowanego biomarkera EC, i PCDHGB7.Surowica CA-125 miała czułość 24,8%, co sugeruje, że CA-125 jest niewystarczającym markerem EC pomimo swoistości 92,3%31.Wykrywanie PCDHGB7 przy użyciu próbek pędzla Pap dało czułość 80,65% i swoistość 82,81% dla stadiów ECatall, podczas gdy szczoteczka Tao miała czułość 61,29% i swoistość 95,31%31.Model diagnostyczny PCDHGB7, oparty na Me-qPCR, dał czułość 98,61%, swoistość 60,5% i ogólną dokładność 85,5% przy użyciu próbek pędzli Pap i Tao (ryc. 4C)31.
Rak urotelialny
Rak urotelialny, obejmujący raka pęcherza moczowego, miedniczki nerkowej i moczowodu, był siódmym najczęściej rozpoznawanym nowotworem w 2020 roku na świecie, powodując 5,2% nowych przypadków i 3,9% zgonów1.Raki nabłonka dróg moczowych, z których ponad 50% to rak pęcherza moczowego, były czwartym najczęściej diagnozowanym nowotworem w Stanach Zjednoczonych w 2022 r. i stanowiły 11,6% nowo zdiagnozowanych przypadków3.Około 75% nowotworów pęcherza moczowego klasyfikuje się jako raka pęcherza moczowego nienaciekającego mięśnia, ograniczonego do błony śluzowej lub podśluzowej45.Biopsja cystoskopowa jest złotym standardem w diagnostyce raka urotelialnego, realizowaną za pomocą fluorescencyjnej hybrydyzacji in situ (FISH) i testów cytologicznych.FISH i cytologia mają słabą skuteczność diagnostyczną, a cystoskopia jest inwazyjna i niesie ze sobą ryzyko przeoczenia mikrouszkodzeń, błędnej interpretacji zmian i potencjalnego spowodowania rozprzestrzeniania się lub nawrotu nowotworu46.Wykazano również, że wcześniej zatwierdzony UCOM, PCDHGB7, jest hipermetylowany w raku nabłonka dróg moczowych, a pole pod krzywą wynosi 0,86, co sugeruje potencjalną zdolność diagnostyczną30.W celu dalszej walidacji większej liczby UCOM i lepszego dostosowania do większej liczby typów próbek zbadano nowatorski UCOM SIX6, który wykazał doskonały potencjał diagnostyczny we wczesnym wykrywaniu raka nabłonka dróg moczowych przy użyciu próbek moczu na platformie Me-qPCR.Wykrywanie SIX6 przy użyciu próbek moczu wykazało czułość konkurencyjną na poziomie 86,7% i swoistość na poziomie 90,8% (ryc. 4D), a jednocześnie było nieinwazyjne i łatwe do uzyskania32.Obecnie badany jest potencjał SIX6 w monitorowaniu przerzutów i ocenie skuteczności leczenia.
Przyszłość i wyzwania
UCOM charakteryzują się dużym potencjałem diagnostycznym wielu nowotworów, pozostaje jednak wiele do zrobienia.Rozszerzamy listę UCOM i aktywnie weryfikujemy UCOM w większej liczbie typów nowotworów, w tym tych, które tradycyjnie są trudne do wykrycia.Wyniki walidacji z baz danych TCGA dodatkowo potwierdziły zastosowanie UCOM w większej liczbie typów nowotworów i większej liczbie sytuacji.We wstępnym badaniu wykazano, że UCOM mają duży potencjał diagnostyczny w przypadku raków dróg żółciowych i gruczolakoraka trzustki, których rozpoznanie na wczesnym etapie przy użyciu obecnych metod przesiewowych jest prawie niemożliwe32,47.Zdolność do wykrywania rzadkich nowotworów za pomocą UCOM można wykorzystać w przypadku krążącego DNA guza (ctDNA) za pomocą ulepszonej platformy do biopsji płynnej48.Badanie z udziałem panelu wykrywającego raka panraka opartego na DNA osocza dało czułość 57,9%49.Pomimo wysokiej specyficzności, ogólna wydajność pokazuje, że nadal jest miejsce na ulepszenia.
Unikalne cechy UCOM wsparły również badanie potencjału UCOM w ocenie skuteczności leczenia i monitorowaniu nawrotów.Zgodnie z kryteriami oceny reakcji w guzach litych (RECIST) obrazowanie medyczne jest zalecaną metodologią monitorowania nawrotów i oceny skuteczności leczenia, podczas gdy do oceny wykorzystuje się wyłącznie markery nowotworowe50.W rzeczywistości jednak na metody obrazowania duży wpływ ma częstotliwość i czas wykonania badania, dlatego narażają pacjentów na większe ryzyko i koszty51,52.Potwierdzono, że SIX6 może służyć jako czynnik prognostyczny przerzutów raka piersi32.Monitorowanie ctDNA w oparciu o biopsję płynną umożliwia nadzór w czasie rzeczywistym przez kilka miesięcy przed wykryciem choroby resztkowej o minimalnej chorobie, co idealnie opóźnia postęp nowotworu związany z nawrotem choroby i zapobiega mu53.Wstępne wyniki sugerują, że UCOM odzwierciedlają poziom hipermetylacji nowotworowej w czasie rzeczywistym bezpośrednio po operacji i leczeniu32.Wysoka czułość wykazywana przez UCOM i możliwość zastosowania w wielu nieinwazyjnych typach próbek pozwala UCOM służyć jako precyzyjny biomarker monitorujący nawroty, przy jednoczesnym zachowaniu wysokiego poziomu przestrzegania zaleceń pacjenta.
Jednocześnie publiczna dostępność testu to kolejna ważna kwestia wymagająca dodatkowego wysiłku.Chociaż w większej liczbie szpitali podjęto współpracę w zakresie wykrywania zakażeń UCOM w nadziei, że przyniesie korzyści większej liczbie pacjentów, na obszarach wiejskich Chin aktywnie przeprowadzano wykrywanie i badania pro bono.UCOM wymagają lepszej dostępności, aby kwalifikować się jako wykonalne narzędzie kontroli, szczególnie w przypadku obszarów słabo rozwiniętych.
Chociaż wyniki zastosowania UCOM we wczesnym wykrywaniu są obiecujące, istnieje wiele niewiadomych na temat UCOM.Dzięki aktywnym eksploracjom uzasadnione są dodatkowe badania nad tym, dlaczego UCOM są powszechnie obecne w nowotworach.Podstawowe mechanizmy regulacji epigenetycznej leżące u podstaw UCOM zasługują na dalsze badania, które mogą uzasadniać nowy kierunek terapii przeciwnowotworowej.Wracając do wzajemnego oddziaływania homogeniczności i heterogeniczności nowotworu, interesuje nas, dlaczego UCOM może stanowić wyjątek od większości biomarkerów nowotworowych, które są ściśle powiązane z określonymi typami nowotworów.Rola zidentyfikowanych przez UCOM aberracji metylacji DNA w powstawaniu nowotworów, progresji nowotworu i przerzutach nie została ustalona w procesie utraty i odzyskiwania tożsamości komórki i wymaga dokładnej kontroli.Kolejnym dużym zainteresowaniem jest zakres włączenia cechy jednorodności UCOM do markerów unikalnych dla tkanki w nadziei na odwrotne podejście do precyzyjnego wykrywania śladów nowotworu i identyfikacji pochodzenia tkanki nowotworowej.UCOM mogą być idealnym narzędziem do zapobiegania nowotworom, wykrywania nowotworów oraz potencjalnej obrony i eliminowania nowotworów.
Udziel wsparcia
Prace te były wspierane przez Chiński Narodowy Program Badań i Rozwoju (grant nr 2022BEG01003), Chińską Narodową Fundację Nauk Przyrodniczych (grant nr 32270645 i 32000505), grant Komisji Zdrowia prowincji Heilongjiang (grant nr 2020-111) oraz grant Instytutu Nauki i Technologii Heze (grant nr 2021KJPT07).
Oświadczenie o konflikcie interesów
Wei Li jest dyrektorem ds. badań i rozwoju w Shanghai Epiprobe Biotechnology Co., Ltd. Wenqiang Yu zasiada w Naukowej Radzie Doradczej Epiprobe.W. Yu i Epiprobe zatwierdzili oczekujące patenty związane z tą pracą.Pozostali autorzy nie deklarują żadnych konkurencyjnych interesów.
Autorskie Wkłady
Projekt wymyślili i zaprojektowali: Chengchen Qian i Wenqiang Yu.
Napisał artykuł: Chengchen Qian.
Wykonał ilustracje: Chengchen Qian.
Recenzowali i redagowali manuskrypt: Xiaolong Zou, Wei Li, Yinshan Li i Wenqiang Yu.
Bibliografia
1. Sung H, Ferlay J, Siegel RL, LaversanneM, Soerjomataram I, Jemal A i in.Globalne statystyki dotyczące raka 2020: szacunki GLOBOCAN dot
zapadalność i śmiertelność na całym świecie w przypadku 36 nowotworów w 185 krajach.CA Rak J Clin.2021;71: 209-49.
2. Xia C, Dong X, Li H, Cao M, Sun D, He S i in.Statystyki nowotworów w Chinach i Stanach Zjednoczonych, 2022: profile, trendy i determinanty.Chin MedJ (angielski).2022;135: 584-90.
3. Siegel RL, Miller KD, WagleNS, JemalA.Statystyki dotyczące raka, 2023. CA Cancer J Clin.2023;73: 17-48.
4. Crosby D, BhatiaS, Brindle KM, Coussens LM, Dive C, Emberton M i in.Wczesne wykrywanie raka.Nauka.2022;375: eaay9040.
5. Ladabaum U, Dominitz JA, KahiC, Schoen RE.Strategie dla
badania przesiewowe w kierunku raka jelita grubego.Gastroenterologia.2020;158: 418-32.
6. Tanoue LT, Tanner NT, Gould MK, Silvestri GA.Badania przesiewowe w kierunku raka płuc.Am J Respir Crit Care Med.2015;191: 19-33.
7. Bouvard V, WentzensenN, Mackie A, Berkhof J, BrothertonJ, Giorgi-Rossi P i in.Perspektywa IARC w zakresie badań przesiewowych w kierunku raka szyjki macicy.N EnglJ Med.2021;385: 1908-18.
8. Xue P, Ng MTA, QiaoY.Wyzwania kolposkopii w badaniach przesiewowych raka szyjki macicy w LMIC i rozwiązaniach wykorzystujących sztuczną inteligencję.BMC Med.2020;18:169.
9. Johnson P, Zhou Q, Dao DY, Lo YMD.Krążące biomarkery w diagnostyce i leczeniu raka wątrobowokomórkowego.Nat Rev Gastroenterol Hepatol.2022;19: 670-81.
10. Van PoppelH, Albreht T, Basu P, HogenhoutR, CollenS, Roobol M. Surowica Wczesne wykrywanie raka prostaty w oparciu o PSA w Europie i na świecie: przeszłość, teraźniejszość i przyszłość.Nat ks Urol.2022;19:
562-72.
11. HolyoakeA, O'Sullivan P, Pollock R, Best T, Watanabe J, KajitaY,
i in.Opracowanie multipleksowego testu moczu RNA do wykrywania i stratyfikacji raka przejściowokomórkowego pęcherza moczowego.Clin Cancer Res.2008;14:742-9.
12. Feinberg AP, Vogelstein B. Hipometylacja odróżnia geny niektórych ludzkich nowotworów od ich normalnych odpowiedników.Natura.1983;301: 89-92.
13. Ng JM, Yu J. Promotor hipermetylacji genów supresorowych nowotworów jako potencjalne biomarkery w raku jelita grubego.IntJ Mol Sci.2015;16:2472-96.
14. Esteller M. Epigenomika nowotworów: metylomy DNA i mapy modyfikacji histonów.Nat Rev Genet.2007;8:286-98.
15. Nishiyama A, Nakanishi M. Nawigacja po metylacji DNA nowotworów.Trendy Genet.2021;37: 1012-27.
16. Xie W, Schultz MD, ListerR, Hou Z, Rajagopal N, Ray P i in.Analiza epigenomiczna wieloliniowego różnicowania ludzkich embrionalnych komórek macierzystych.Komórka.2013;153: 1134-48.
17. Li Y, Zheng H, Wang Q, Zhou C, WeiL, Liu X i in.Analizy obejmujące cały genom ujawniają rolę Polycombu w promowaniu hipometylacji dolin metylacji DNA.Biol genomu.2018;19:18.
18. Koch A, JoostenSC, Feng Z, de Ruijter TC, DrahtMX, MelotteV,
i in.Analiza metylacji DNA w raku: ponowna lokalizacja.Nat Rev Clin Oncol.2018;15: 459-66.
19. KleinEA, Richards D, Cohn A, TummalaM, Lapham R, Cosgrove D i in.Walidacja kliniczna ukierunkowanego testu wczesnego wykrywania wielu nowotworów opartego na metylacji przy użyciu niezależnego zestawu walidacyjnego.Anna Onkol.2021;32: 1167-77.
20. Hanahan D, Weinberg RA.Cechy charakterystyczne raka.Komórka.2000;100: 57-70.
21. Hanahan D. Cechy charakterystyczne raka: nowe wymiary.Dyskusja na temat raka2022;12:31-46.
22. Schwartzberg L, Kim ES, Liu D, Schrag D. Onkologia precyzyjna: kto, jak, co, kiedy i kiedy nie?Książka Am Soc Clin Oncol Educ.2017: 160-9.
23. Liu H, Meng X, Wang J. Ilościowa metylacja w czasie rzeczywistym
wykrywanie genu PAX1 w badaniach przesiewowych w kierunku raka szyjki macicy.IntJ Gynecol Rak.2020;30: 1488-92.
24. Imperiale TF, RansohoffDF, Itzkowitz SH, Levin TR, Lavin P, Lidgard GP i in.Wielodocelowe badanie DNA kału do badań przesiewowych w kierunku raka jelita grubego.N EnglJ Med.2014;370: 1287-97.
25. Li J, Li Y, Li W, Luo H, Xi Y, Dong S i in.Pozycjonowanie przewodnika
sekwencjonowanie identyfikuje nieprawidłowe wzorce metylacji DNA, które zmieniają tożsamość komórek i sieci nadzoru odporności na nowotwory.Genom
Rozdzielczość2019;29: 270-80.
26. Gao Q, LinYP, Li BS, Wang GQ, Dong LQ, Shen BY i in.Nieinwazyjne wykrywanie wielu nowotworów za pomocą sekwencjonowania metylacji krążącego bezkomórkowego DNA (THUNDER): rozwój i niezależne badania walidacyjne.Anna Onkol.2023;34: 486-95.
27. Dong S, Li W, Wang L, Hu J, Song Y, Zhang B i in.Geny związane z histonami są hipermetylowane w raku płuc i hipermetylowane
HIST1H4F może służyć jako biomarker panrakowy.Rak Res.2019;79: 6101-12.
28. HeijnsdijkEA, Wever EM, AuvinenA, Hugosson J, Ciatto S, Nelen V i in.Wpływ na jakość życia badań przesiewowych pod kątem antygenów specyficznych dla prostaty.N EnglJ Med.2012;367: 595-605.
29. LuzakA, Schnell-Inderst P, Bühn S, Mayer-Zitarosa A, Siebert U. Skuteczność kliniczna testów biomarkerów do badań przesiewowych w kierunku raka oferowanych w ramach samopłatnej usługi zdrowotnej: przegląd systematyczny.Eur J. Zdrowie publiczne.2016;26: 498-505.
30. Dong S, Lu Q, Xu P, Chen L, Duan X, Mao Z i in.
Hipermetylowany PCDHGB7 jako uniwersalny marker wyłącznie nowotworowy i jego zastosowanie we wczesnych badaniach przesiewowych w kierunku raka szyjki macicy.Clin Transl Med.2021;11: e457.
31. Yuan J, Mao Z, Lu Q, Xu P, Wang C, Xu X i in.Hipermetylowany PCDHGB7 jako biomarker do wczesnego wykrywania raka endometrium w próbkach szczoteczki endometrium i zeskrobinach szyjki macicy.Front Mol Biosci.2022;8: 774215.
32. Dong S, Yang Z, Xu P, Zheng W, Zhang B, Fu F i in.Wzajemnie
Ekskluzywna modyfikacja epigenetyczna SIX6 z hipermetylacją w celu śledzenia stanu przedrakowego i pojawiania się przerzutów.Cel przekazywania sygnału Tam.2022;7: 208.
33. Huang L, Guo Z, Wang F, Fu L. Mutacja KRAS: od nieuleczalnej do narkotykowej w przypadku raka.Cel przekazywania sygnału Tam.2021;6: 386.
34. Belinsky SA, Nikula KJ, PalmisanOWA, MichelsR, SaccomannoG, GabrielsonE i in.Nieprawidłowa metylacja p16(INK4a) jest zjawiskiem występującym na wczesnym etapie raka płuc i potencjalnym biomarkerem do wczesnej diagnostyki.Proc Natl Acad Sci U SA.1998;95: 11891-6.
35. Robertson KD.Metylacja DNA i choroby człowieka.Nat Rev Genet.2005;6: 597-610.
36. WentzensenN, Walker JL, Gold MA, Smith KM, ZunaRE,
Mathews C i in.Biopsje wielokrotne i wykrywanie prekursorów raka szyjki macicy podczas kolposkopii.J Clin Oncol.2015;33: 83-9.
37. De Strooper LM, Meijer CJ, Berkhof J, Hesselink AT, Snijders
PJ, Steenbergen RD i in.Analiza metylacji FAM19A4
gen w zadrapaniach szyjki macicy jest wysoce skuteczny w wykrywaniu szyjki macicy
nowotwory i zaawansowane zmiany CIN2/3.Rak Poprzedni Res (Phila).2014;7: 1251-7.
38. Thai AA, Solomon BJ, Sequist LV, Gainor JF, Heist RS.Rak płuc.Lancet.2021;398: 535-54.
39. Grunnet M, Sorensen JB.Antygen rakowo-embrionalny (CEA) jako marker nowotworowy w raku płuc.Rak płuc.2012;76: 138-43.
40. Wood DE, KazerooniEA, Baum SL, EapenGA, EttingerDS, Hou L i in.Badania przesiewowe w kierunku raka płuc, wersja 3.2018, Wytyczne praktyki klinicznej NCCN w onkologii.J Natl Compr Canc Netw.2018;16: 412-41.
41. Amerykańskie Towarzystwo Onkologiczne.Fakty i liczby dotyczące raka.Atlanta, Georgia, USA: Amerykańskie Towarzystwo ds. Walki z Rakiem;2023 [aktualizacja 2023 1 marca;cytowano 22 sierpnia 2023 r.].
42. FonthamETH, Wolf AMD, Church TR, EtzioniR, Flowers CR,
Herzig A i in.Badania przesiewowe w kierunku raka szyjki macicy u osób o średnim ryzyku: aktualizacja wytycznych American Cancer Society na rok 2020.CA Rak J Clin.2020;70: 321-46.
43. Clarke MA, Long BJ, Del Mar MorilloA, Arbyn M, Bakkum-Gamez JN, Wentzensen N. Związek ryzyka raka endometrium z krwawieniem pomenopauzalnym u kobiet: przegląd systematyczny i metaanaliza.JAMA Stażysta Med.2018;178: 1210-22.
44. Jacobs I, Gentry-MaharajA, Burnell M, ManchandaR, Singh N,
Sharma A i in.Czułość przezpochwowego badania USG
w przypadku raka endometrium u kobiet po menopauzie: badanie kliniczno-kontrolne w kohorcie UKCTOCS.Lancet Onkol.2011;12:38-48.
45. BabjukM, Burger M, CompératEM, Gontero P, MostafidAH,
PalouJ i in.Wytyczne Europejskiego Towarzystwa Urologicznego dotyczące nieinwazyjnego raka pęcherza moczowego (TaT1 i rak in situ) -
Aktualizacja 2019.EUR Urol.2019;76: 639-57.
46. Aragon-Ching JB.Wyzwania i postępy w diagnostyce, biologii i leczeniu raka górnych dróg moczowych i raka pęcherza moczowego.Urol Onkol.2017;35: 462-4.
47. Rizvi S, KhanSA, Hallemeier CL, Kelley RK, Gores GJ.
Rak dróg żółciowych – ewoluujące koncepcje i strategie terapeutyczne.Nat Rev Clin Oncol.2018;15: 95-111.
48. Ye Q, Ling S, Zheng S, Xu X. Biopsja płynna w komórkach wątrobowych
rak: krążące komórki nowotworowe i krążący DNA nowotworu.Mol Rak.2019;18:114.
49. Zhang Y, Yao Y, Xu Y, Li L, Gong Y, Zhang K i in.Panrak
wykrycie krążącego DNA nowotworu u ponad 10 000 chińskich pacjentów.Nat Commun.2021;12:11.
50. Eisenhauer EA, Therasse P, BogaertsJ, Schwartz LH, Sargent D, Ford R i in.Nowe kryteria oceny odpowiedzi w guzach litych: zmienione wytyczne RECIST (wersja 1.1).Rak Eur J.2009;45:228-47.
51. LitièreS, Collette S, de Vries EG, Seymour L, BogaertsJ.RECIST – nauka z przeszłości, aby budować przyszłość.Nat Rev Clin Oncol.
2017;14: 187-92.
52. Seymour L, BogaertsJ, Perrone A, FordR, Schwartz LH, Mandrekar S i in.iRECIST: wytyczne dotyczące kryteriów odpowiedzi do stosowania w badaniach
testowanie leków immunoterapeutycznych.Lancet Onkol.2017;18: e143-52.
53. PantelK, Alix-Panabières C. Biopsja płynna i minimalna choroba resztkowa – najnowsze osiągnięcia i implikacje w zakresie leczenia.Nat Rev Clin Oncol.2019;16:409-24.
Cytuj ten artykuł jako: Qian C, Zou X, Li W, Li Y, Yu W. Placówka przeciwko rakowi: uniwersalne markery tylko dla raka.Rak Biol Med.2023;20: 806-815.
doi: 10.20892/j.issn.2095-3941.2023.0313
Czas publikacji: 7 maja 2024 r